はじめに
GLB事業部Lakehouse部の長尾です。
本ブログ(と後編)では、Databricks社の Training machine learning models on tabular data: an end-to-end example (Unity Catalog) を参照して(2025年1月16日時点)、MLflow & Hyperoptを活用したDatabricks上で機械学習を効率化させるための実装・管理方法について紹介します。
ひとつのパターンとして本ブログでの一連の実行の流れを覚えておけると他のケースでも応用できるのではないかと考えています。
※本ブログは上記参照先の内容の翻訳ではありませんが内容はほぼ同じです。
参照先には記載がない内容も補足説明として多少追加しています。
Databricks上での機械学習を効率化させるための実装方法について、主に以下の2つの観点から説明します。
- DatabricksのUnity Catalogを活用して、どのように機械学習モデルを訓練して登録(Unity Catalogの中でMLflowを活用した再現性の強化)
- Hyperoptを活用してハイパーパラメータを効率的にチューニング ※文字数の都合上、Hyperoptについては後編で説明しています。
以下のステップで実装します。 (以下の②~④については後編で説明しています。)
- ① seabornやmatplotlibを使ったデータの可視化
- ② 並列でハイパーパラメータの最適な組み合わせを探索・試行し複数のモデルをトレーニング
- ③ MLflowを使用してハイパーパラメータの探索・試行の結果を調査
- ④ 登録済みのモデルをSpark UDFを使用して別のデータセットに適用(新規のデータセットに対してMLflowに登録済みの機械学習モデルをすぐに利用して予測結果を生成するため)
今回の例では、ポルトガルのワイン「Vinho Verde」の品質を物理化学的特性に基づいて予測するモデルを構築します。
【以降の内容を実施するための必須条件】
- Databricks上で以降の内容を実行するためには、Databricks Runtime 15.4 LTS ML以上が動作しているクラスターが必要です。
- Unity Catalogが有効になっているワークスペースが必要です(Unity Catalogが有効になってない場合の実行方法はこちら)
それでは、以降で実際に実行してみましょう。
Unity Catalogのセットアップ まずは、mlflowをインストールして、Unity Catalogのセットアップします。
- モデルを登録するカタログとスキーマを設定します。
- 使用するカタログに対するUSE CATALOG権限、またスキーマに対するCREATE MODEL権限とUSE SCHEMA権限が必要です。
%pip install mlflow import mlflow mlflow.set_registry_uri("databricks-uc") CATALOG_NAME = "main" SCHEMA_NAME = "default"
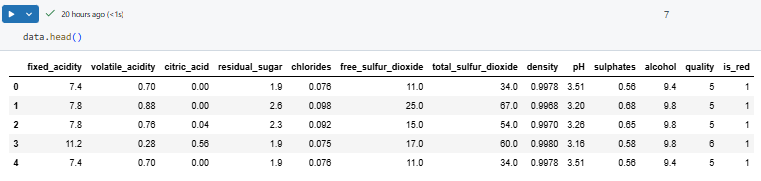
使用するデータの読み込み 白ワインと赤ワインの品質に関するCSVデータセットを読み込みます。
import pandas as pd white_wine = pd.read_csv("/databricks-datasets/wine-quality/winequality-white.csv", sep=";") red_wine = pd.read_csv("/databricks-datasets/wine-quality/winequality-red.csv", sep=";")
読みんだ2つのデータをマージして1つのデータフレームにした後、赤ワインか白ワインかを示す2値データを「is_red」列として新しく追加します。
red_wine['is_red'] = 1 white_wine['is_red'] = 0 data = pd.concat([red_wine, white_wine], axis=0) # 列名からスペースを削除 data.rename(columns=lambda x: x.replace(" ", "_"), inplace=True)
意図しているようになっているか、データの中身を確認しておきます。
data.head()
data.isna().sum()
 欠損値はありません。
欠損値はありません。
データを可視化 モデルをトレーニングする前に、SeabornとMatplotlibを使ってデータセットを確認してみましょう。 目的変数であるqualityデータの分布をヒストグラムで確認します。
import seaborn as sns sns.displot(data.quality, kde=False)
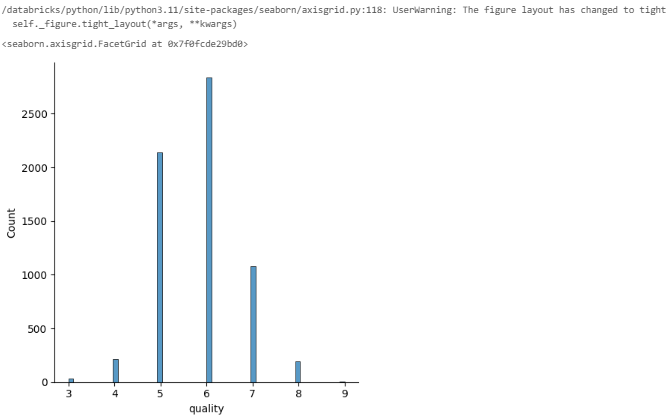 qualityは3~9の間で正規分布しているように見えます。
ここでは、もしqualityが7以上であれば高品質であると定義します。
qualityは3~9の間で正規分布しているように見えます。
ここでは、もしqualityが7以上であれば高品質であると定義します。
high_quality = (data.quality >= 7).astype(int) data.quality = high_quality
箱ひげ図は、各データの分布、特徴量と目的変数(quality)の関係性、外れ値等を確認する際に役立ちます。 low-qualityとhigh-qualityで特徴量にどのような違いがあるのかを可視化します。 もしlow-qualityとhigh-qualityで特徴量の分布に明らかな違いがあれば、その特徴量が品質を予測する有用な説明変数である可能性があります。
import matplotlib.pyplot as plt dims = (3, 4) f, axes = plt.subplots(dims[0], dims[1], figsize=(25, 15)) axis_i, axis_j = 0, 0 for col in data.columns: if col == 'is_red' or col == 'quality': continue sns.boxplot(x=high_quality, y=data[col], ax=axes[axis_i, axis_j]) axis_j += 1 if axis_j == dims[1]: axis_i += 1 axis_j = 0
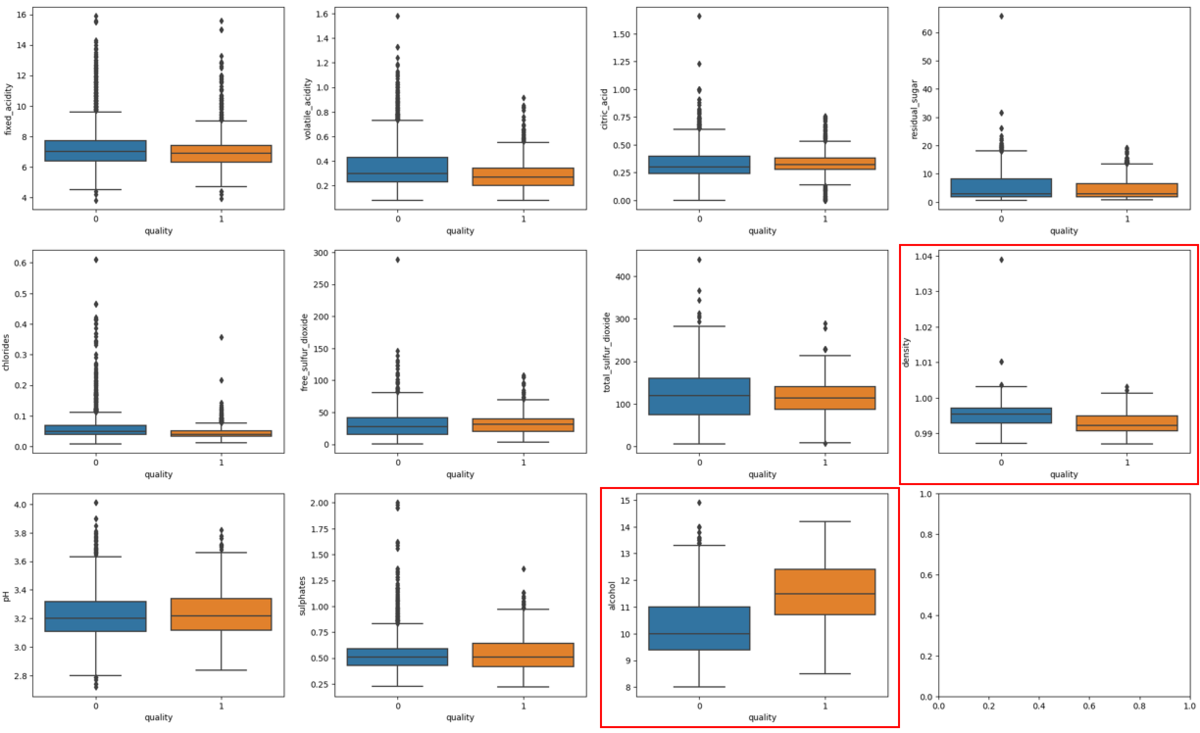 <上記の箱ひげ図から視覚的に分かること>
<上記の箱ひげ図から視覚的に分かること>
- 箱ひげ図のalcohol(3段目の赤枠)は、high-qualityの中央値がlow-qualityの75パーセント点(第3四分位点)よりも大きいです。 このことから、alcoholの濃度はqualityと関係性がありそうです。
- density(2段目の赤枠)は、low-qualityの方がlow-qualityよりもdensityが高いようです。 そのため、densityはqualityと負の相関があると考えられます。
赤ワインと白ワインのサンプルサイズの分布とそれぞれで高品質がどのくらいあるのかが気になったので、クロス集計表を作成して確認します。
crosstab_table = pd.crosstab(data.is_red, data.quality, rownames=['is_red'], colnames=['data_quality']) # 行ごとの割合を計算 crosstab_percentage = crosstab_table.div(crosstab_table.sum(axis=1), axis=0)*100 # Crosstabに割合を追加 crosstab_with_percentage = crosstab_table.copy() crosstab_with_percentage["% Low Quality"] = crosstab_percentage[0] # Low Qualityの割合 crosstab_with_percentage["% High Quality"] = crosstab_percentage[1] # High Quality割合 crosstab_with_percentage['Total'] = crosstab_table.sum(axis=1) # 行合計を追加 print(crosstab_with_percentage)
 今回のサンプル全体の約75%が白ワインで、高品質である割合は白ワインの方が赤ワインよりも約8%高いようです(しかしながら、あとの結果を基にすると赤ワインか白ワインかよりも他の特徴量のほうが予測に寄与しているようです)。
今回のサンプル全体の約75%が白ワインで、高品質である割合は白ワインの方が赤ワインよりも約8%高いようです(しかしながら、あとの結果を基にすると赤ワインか白ワインかよりも他の特徴量のほうが予測に寄与しているようです)。
ベースラインモデルを訓練するためのデータセットを準備
- 訓練データとして60%を使用し、残りの40%を_remとします。
- 次に、_remをテストデータと検証用データに半分ずつ分けます。
from sklearn.model_selection import train_test_split X = data.drop(['quality'], axis=1) y = data.quality # トレーニングデータを分割 X_train, X_rem, y_train, y_rem = train_test_split(X, y, train_size=0.6, random_state=123) # 残りのデータを検証用データとテスト用データに均等に分割 X_val, X_test, y_val, y_test = train_test_split(X_rem, y_rem, train_size=0.5, random_state=123)
ベースラインモデルを訓練
- 2値データであるqualityを目的変数とし、かつ複数の変数間で相互作用も考えられるため、random forest classifierを使用することがふさわしいと考えられます。
- scikit-learnを使用してシンプルな分類器を作成し、MLflowを使ってモデルの精度をトラッキングして、今後の使用のためにモデルを保存しておきましょう。
import mlflow.pyfunc import mlflow.sklearn import numpy as np import sklearn from sklearn.ensemble import RandomForestClassifier from sklearn.metrics import roc_auc_score from mlflow.models.signature import infer_signature from mlflow.utils.environment import _mlflow_conda_env import cloudpickle import time # sklearnのpredictメソッド「**RandomForestClassifier**」は、二値分類(0または1)を返します。 # 以下のコードでは、predict_probaメソッドを使用して、観測値が各クラスに属する確率を返すラッパー関数(SklearnModelWrapper)を作成しています。 class SklearnModelWrapper(mlflow.pyfunc.PythonModel): def __init__(self, model): self.model = model def predict(self, context, model_input): return self.model.predict_proba(model_input)[:,1] # 新しいRunを開始する前に、現在アクティブなRunを終了します。 mlflow.end_run() # mlflow.start_runで、このモデルのパフォーマンスを追跡するための新しいMLflow Runを作成します。 # このコンテキスト内では、mlflow.log_paramによって使用したパラメータを記録し、mlflow.log_metricを使用して精度などのメトリクスを記録します。 with mlflow.start_run(run_name='untuned_random_forest'): n_estimators = 10 model = RandomForestClassifier(n_estimators=n_estimators, random_state=np.random.RandomState(123)) model.fit(X_train, y_train) # predict_proba は [prob_negative, prob_positive](0のときの確率と1のときの確率)を返すため、出力を [:, 1] でスライスして正クラス(1のとき)の確率を取得します。 predictions_test = model.predict_proba(X_test)[:,1] auc_score = roc_auc_score(y_test, predictions_test) mlflow.log_param('n_estimators', n_estimators) # ROC曲線の下の面積(AUC)をメトリクスとして使用します。 mlflow.log_metric('auc', auc_score) wrappedModel = SklearnModelWrapper(model) # モデルの入力と出力のスキーマを定義するシグネチャを使ってモデルを記録します。 # モデルがデプロイされる際に、このシグネチャは入力の検証に使用されます。 signature = infer_signature(X_train, wrappedModel.predict(None, X_train)) # MLflowには、モデルを提供するためのConda環境を作成する機能が含まれています。 # 必要な依存関係は conda.yamlファイルに追加され、モデルと共に記録されます。 conda_env = _mlflow_conda_env( additional_conda_deps=None, additional_pip_deps=["cloudpickle=={}".format(cloudpickle.__version__), "scikit-learn=={}".format(sklearn.__version__)], additional_conda_channels=None, ) mlflow.pyfunc.log_model("random_forest_model", python_model=wrappedModel, conda_env=conda_env, signature=signature)
モデルによって算出されたFeature Importancesを見てみましょう。
feature_importances = pd.DataFrame(model.feature_importances_, index=X_train.columns.tolist(), columns=['importance']) feature_importances.sort_values('importance', ascending=False)
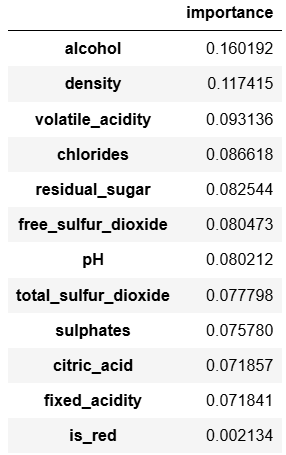
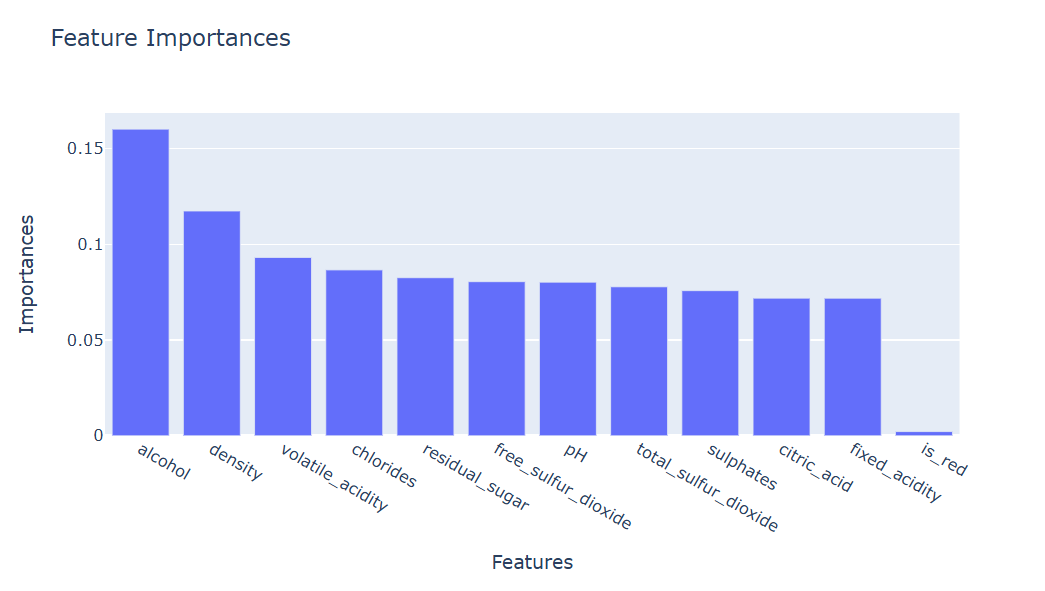 上記の結果からも、箱ひげ図で確認した内容と同様に、qualityを予測する際にalcoholとdensityが重要な特徴量である可能性が高そうです。
上記の結果からも、箱ひげ図で確認した内容と同様に、qualityを予測する際にalcoholとdensityが重要な特徴量である可能性が高そうです。
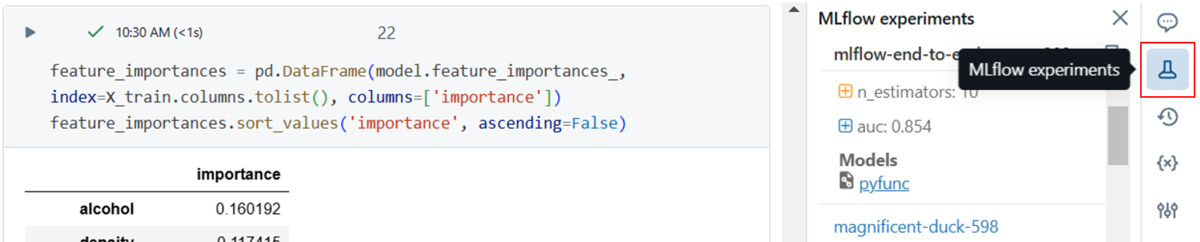
Area Under the ROC Curve (AUC)がMLflowにログされます。
以下のように画面右(赤枠内)のMLflow experimentsをクリックするとAUCを確認できます。
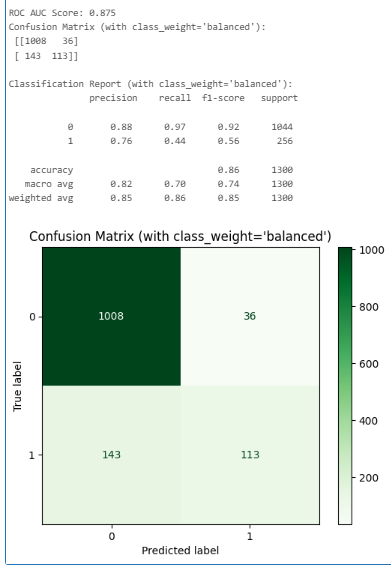
今回は、AUCが0.854です。
(もし完全にランダムな予測であればAUCは0.5、予測が完璧であればAUCは1.0です)
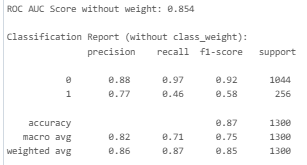
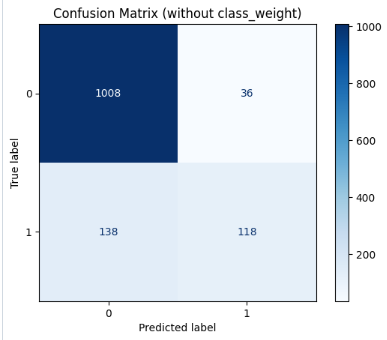
ちなみに、「不均衡なデータセット」(今回は目的変数であるqualityの「1」の割合が約20%)に対処する方法のひとつとして、class_weight='balanced'をRandomForestClassifierのインスタンスを作成する際に引数として指定することができます。
RandomForestClassifierは、bootstrap sampling(データの再サンプリングを繰り返す)を使って各決定木を学習させており、多様なデータセットを基にしつつ汎化性能を向上させる手法です。
そのため、デフォルトでもクラスの不均衡に比較的強いアルゴリズムですが、試しにclass_weight='balanced'と指定してみます。
class_weight='balanced'を使用した場合、AUCは0.875と少し高くなりました(class_weightを指定しない場合は0.854)。
<参考>
- Receiver Operating Characteristic Curve
- Scaling Hyperopt to Tune Machine Learning Models in Python
- Hyperparameter Tuning with MLflow, Apache Spark MLlib and Hyperopt
最後に 本ブログでは、Databricks上でMLflowを活用した効率的な機械学習の実装・管理方法について紹介しました。 文字数の都合上、本ブログで割愛したHyperoptについては後編で説明していますのでご一読いただけますと幸いです。 長文にもかかわらず最後まで読んでいただき、ありがとうございました。
私たちはDatabricksを用いたデータ分析基盤の導入から内製化支援まで幅広く支援をしております。
もしご興味がある方は、お問い合わせ頂ければ幸いです。
また、一緒に働いていただける仲間も募集中です!
APCにご興味がある方の連絡をお待ちしております。